Replicação
📧
- Faculdade de Ciências da Universidade de Lisboa
Referência Moreira, C., (2014) Replicação, Rev. Ciência Elem., V2(1):096
DOI http://doi.org/10.24927/rce2014.096
Palavras-chave Replicação; DNA; hipótese semi-conservativa;
Resumo
A replicação é um processo de síntese de novas cadeias de DNA a partir de cadeias de DNA parentais utilizadas como molde. Nos humanos e outros eucariotas, a replicação ocorre no núcleo da célula.
Baseado no modelo de dupla hélice proposto por Watson e Crick, em 1953, de que resulta a ideia de emparelhamento das bases do DNA (complementaridade das bases) é avançada a hipótese de que o DNA tem a capacidade de se auto-duplicar. Este processo sabe-se hoje ser semiconservativo – hipótese semiconservativa – isto é, cada uma das novas molécula de DNA resultantes de replicação é constituída por uma cadeia proveniente da molécula original de DNA que serviu de molde e por uma cadeia complementar anti-paralela sintetizada de novo.
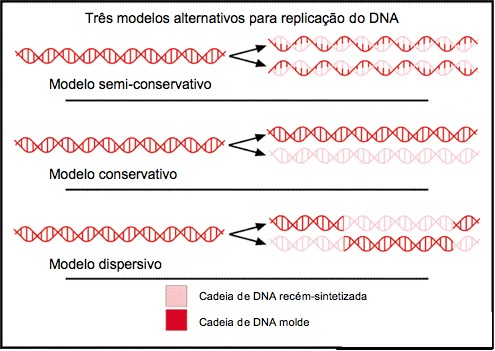
Outros investigadores sugeriram modelos alternativos: a hipótese conservativa admitia que a molécula de DNA original se mantinha, servindo apenas de molde para a formação de uma nova molécula formada por duas novas cadeias de nucleótidos; a hipótese dispersiva, por seu lado, admitia que as novas moléculas de DNA teriam cadeias formadas por porções da molécula inicial e por porções sintetizadas de novo.
Em 1956, Arthur Kornberg demonstrou que era possível replicar a molécula de DNA em laboratório, i.e., no exterior da célula. Para esta replicação in vitro é necessário para além da molécula de DNA parental, a enzima polimerase do DNA e os quatro tipos de nucleótidos (adenina, citosina, timina e guanina) que formam este ácido nucleico.
Embora Watson e Crick tivessem postulado sobre a replicação semiconservativa das cadeias de DNA e de Kornberg ter replicado em laboratório moléculas de DNA, só em 1958 com as experiências de Matthew Meselson e Franklin Stahl (através da marcação do DNA com um isótopo de azoto pesado 15N) se esclareceu o mecanismo de replicação do DNA.
As experiências de Meselson e Stahl
Meselson e Stahl cultivaram bactérias Escherichia coli num meio de cultura enriquecido com um isótopo pesado de azoto 15N, em vez do usual 14N mais abundante e com menor massa. Devido às suas diferentes massas estes dois isótopos podem ser separados por centrifugação, o que permitiria aos investigadores separar moléculas de DNA que incorporassem diferentes isótopos. As bactérias E. coli cresceram durante várias gerações no meio com 15N e após extração do DNA destas células, e centrifugação do mesmo, verificaram que tinha densidade superior ao DNA de bactérias a crescer em meio contendo 14N. Numa segunda fase, bactérias E. coli originalmente a crescer em meio com 15N foram transferidas para um meio com 14N, permitindo-se que realizassem apenas uma divisão celular, com a replicação correspondente do seu DNA. Novamente, o DNA foi extraído e centrifugado. Quando compararam as densidades dos três grupos de DNA extraídos (14N, 15N e 15N - 14N) verificaram que a densidade do DNA das bactérias que tinham crescido nos dois meios (primeiro em 15N e depois 14N) era intermédio do DNA de E. coli cultivado apenas em meios simples de 14N ou 15N (densidade do DNA extraído: 14N DNA < 15N - 14N DNA < 15N DNA). Estes resultados punham de parte a hipótese conservativa de replicação em que se esperaria metade do DNA com densidade equivalente à do 14N e a outra metade com densidade equivalente à do 15N, no entanto, quer a hipótese dispersiva quer a semiconservativa poderiam ser explicadas à luz destes resultados. Se a replicação fosse semiconservativa esperar-se-iam cadeias duplas de DNA com uma cadeia 15N e outra 14N, e se fosse dispersiva ambas as cadeias de DNA teriam uma mistura de 14N e 15N, apresentando nos dois casos densidades intermédias com valores semelhantes.
Para averiguar qual das duas hipóteses explicava a replicação do DNA os dois investigadores extraíram DNA de colónias de E. coli que tinham crescido durante várias gerações em meio com 15N e durante apenas duas divisões em meio com 14N. O DNA extraído consistia em duas porções equivalentes mas de densidades diferentes. Uma correspondia às células que se tinham dividido nos dois meios e, por isso, com uma densidade intermédia e a outra com menor densidade, correspondia às células que se tinham dividido exclusivamente no meio com 14N. Estes segundos resultados eliminariam a hipótese dispersiva onde se esperaria apenas um único grupo de DNA com a mesma densidade, onde as cadeias formadas teriam várias porções de ambos os isótopos.
Como se processa a replicação do DNA
Depois de se identificar o modelo de replicação do DNA, o passo seguinte foi investigar como decorre o processo. Cada cadeia de DNA parental servirá de molde à formação de uma nova cadeia complementar (rever regra de complementaridade das bases azotadas) utilizando os nucleótidos livres no nucleoplasma de cada célula. O resultado final são duas novas moléculas de DNA de cadeia dupla idênticas entre si, com uma cadeia original e outra complementar recém sintetizada.
O processo de replicação dá-se de forma bidirecional, isto é, para ambos os lados da origem, sendo as duas cadeias de DNA sintetizadas em simultâneo. As cadeias complementares são sintetizadas em direção antiparalela à da cadeia molde, no sentido 5’ – 3’ e de forma semi-descontínua, i.e., as cadeias filhas não se formam da mesma maneira: uma cresce de modo contínuo, a cadeia líder (do inglês leading strand) que é complementar à cadeia 3’ – 5’, e a outra cresce pela adição de fragamentos, a cadeia atrasada (do inglês lagging strand), que é complementar à cadeia 5’ – 3’ (ver esquema).
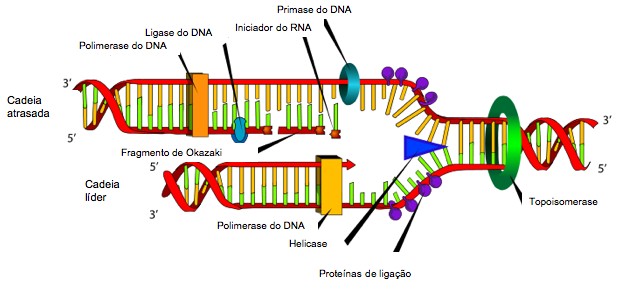
O processo de replicação envolve a participação de várias enzimas, que possibilitam o desenrolar da dupla hélice da molécula de DNA, a separação das suas cadeias e a construção das novas cadeias. Entre elas podemos destacar as polimerases do DNA e as ligases do DNA.
As polimerases do DNA são enzimas responsáveis:
- pela formação de ligações por pontes de hidrogénio entre as bases complementares (A com T e G com C),
- pela ligação do açúcar de um nucleótido com o fosfato do nucleótido seguinte – extremidade 3’ da pentose,
- pela correção de erros na sequência nucleotídica.
As ligases do DNA são responsáveis por:
- ligar os vários fragmentos – fragmentos de Okazaki, que se formam de modo descontínuo na cadeia filha complementar à cadeia parental 5’ - 3’.
A replicação desenrola-se em 3 etapas: iniciação, elongação e terminação.
O mecanismo de replicação, embora comum a todos os organismos, sejam eles eucariotas ou procariotas, apresenta grandes diferenças no processo. Nos procariotas, a replicação inicia-se num único ponto da cadeia polinucleotídica e prossegue até terminar, porque nestes organismos apenas existe uma molécula de DNA e o seu comprimento é muito menor que o do DNA eucariota.
Nas células eucariotas a replicação do DNA ocorre, em simultâneo, em múltiplos locais específicos da cromatina (origens de replicação, são sequências ricas em A-T), catalisada pelas polimerases do DNA. A molécula de DNA, começa a desenrolar por ação das enzimas topoisomerases e das helicases, e a desnaturar ao nível das origens de replicação. Para as polimerases do DNA iniciarem a condensação dos nucleótidos percursores (de acordo com a sequência nucleotídica da cadeia simples de DNA molde) é necessária a síntese de um pequeno fragmento de RNA (designado por iniciador, primer) catalisada por uma polimerase do RNA específica, a primase (uma subunidade da polimerase do DNA a).
A polimerização das novas cadeias de DNA ocorre de forma bidirecional, a partir de cada origem de replicação, formando-se a forquilha de replicação (do inglês ‘replication fork’, ver figura) que se deslocam em direções opostas. Em cada braço da forquilha de replicação a polimerização ocorre no sentido 5’-P – 3’-OH, na cadeia líder de forma contínua e na cadeia atrasada de forma descontínua, com a formação de fragmentos, os fragmentos de Okazaki, a partir de iniciadores de RNA. Posteriormente, os segmentos de iniciadores de RNA são removidos e uma polimerase do DNA sintetiza em seguida DNA que preenche as regiões inicialmente ocupadas pelos iniciadores. A continuidade das cadeias recém sintetizadas é assegurada por uma ligase do DNA que catalisa a formação de ligações fosfodiéster entre os vários fragmentos.
Como se evitam os erros na síntese das novas cadeias
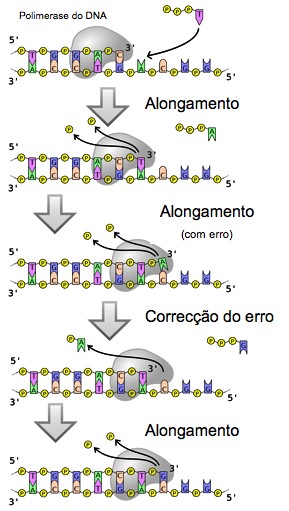
Para evitar que erros que possam ter ocorrido durante a síntese permaneçam nas novas cadeias de DNA replicadas, existe uma auto-correção ao longo do processo. A primeira verificação ocorre antes de cada novo nucleótido ser adicionado à cadeia. Cada nucleótido correto tem maior afinidade com a polimerase do DNA que um nucleótido incorreto, uma vez que apenas o correto emparelha no par da base complementar. Depois da adição de um nucleótido, e antes de este formar ligações covalentes com a cadeia de DNA em formação, a enzima sofre uma alteração conformacional. Mais uma vez a presença do nucleótido errado será possivelmente detetada e o mesmo dissocia-se da cadeia. Pode, no entanto, acontecer que o nucleótido incorreto passe esta primeira verificação mas neste caso a polimerase do DNA é impossibilitada de prosseguir. Num destes raros casos o nucleótido inserido incorretamente é retirado da cadeia pela atividade exonucleásica da polimerase do DNA 3’ – 5’. Esta capacidade de auto-correção permite que não ocorram erros durante o processo de replicação, assegurando a perfeita complementaridade das bases.
Materiais relacionados disponíveis na Casa das Ciências
:
- Código
da Vida – Capítulo 3, o que é o um gene? Como é que um gene
origina uma proteína?
- Tradução
do mRNA, veja como o mRNA se traduz numa proteína
- Splicing
do mRNA, o que acontece ao mRNA antes de poder ser traduzido
numa proteína
- Processamento
do mRNA, o que acontece ao mRNA logo após a transcrição
- Transcrição
do DNA, a transcrição do DNA em mRNA passo a passo
- Dogma
Central do ADN - Parte 2 :Tradução, tradução do ARN
- Dogma
Central do ADN – Parte 1: Transcrição, veja como o ADN é
transcrito no núcleo
- Visualização
Molecular do ADN, veja o enrolamento e a replicação do ADN
- Síntese Proteica - Tradução, veja este processo num flash simples
Este artigo já foi visualizado 20525 vezes.



