Enzima de restrição
📧
- Faculdade de Ciências da Universidade de Lisboa
Referência Moreira, C., (2014) Enzima de restrição, Rev. Ciência Elem., V2(2):033
DOI http://doi.org/10.24927/rce2014.033
Palavras-chave Enzima; infeção; virus,
Resumo
As enzimas de restrição (ou endonucleases de restrição) são enzimas que cortam o DNA em locais específicos. As enzimas reconhecem determinadas sequencias nucleotídicas do DNA e fragmentam a molécula sempre que identificam essa sequência, produzindo extremidades coesivas.
As enzimas de restrição foram descobertas em bactérias que resistiam à infeção dos vírus (bacteriófagos) produzindo enzimas que seccionavam o DNA viral, fragmentando-o em porções inofensivas.
As enzimas atuam quando identificam determinadas sequencias especificas – locais de restrição – geralmente compostas por 4-6 nucleótidos. As enzimas cortam as ligações entre o grupo hidroxilo 3’ de um nucleótido e o grupo fosfato 5’ do nucleótido adjacente. As extremidades das cadeias seccionadas – extremidades coesivas – quando contactam com outras resultantes da ação da mesma enzima podem emparelhar por complementaridade.
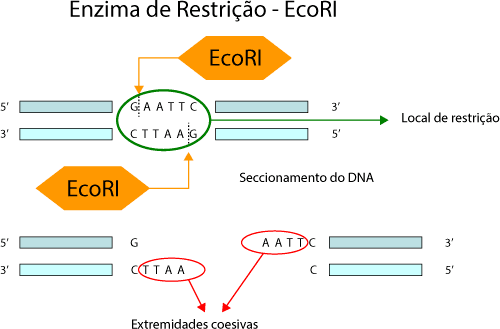
Já foram identificadas várias enzimas de restrição.
A tabela seguinte mostra alguns exemplos de enzimas de restrição, da sequências que reconhecem e a bactéria onde a encontraram.
| Enzima | Bactéria de origem | Sequência de reconhecimento |
| EcoRI | Escherichia coli | 5'GAATTC 3'CTTAAG |
| BamHI | Bacillus amyloliquefaciens | 5'GGATCC 3'CCTAGG |
| TaqI | hermus aquaticus | 5'TCGA 3'AGCT |
| Xbal | Xanthomonas badrii | 5'TCTAGA 3'AGATCT |
Materiais relacionados disponíveis na Casa das Ciências:
- As bactérias E. coli patogénicas e não patogénicas, de Bio-DiTRL;
- Laboratório Virtual de Biotecnologia, de Nuno Ribeiro;
- Extração do DNA, de Diana Lobo;
- Enzima de restrição EcoR1, de Drew Berry.
Este artigo já foi visualizado 6456 vezes.



