O vírus da Gripe
📧 , 📧
- * cE3c - Centro de Ecologia, Evolução e Alterações Ambientais - Universidade de Lisboa
- ɫ Faculdade de Ciências da Universidade de Lisboa
Referência Nogueira, T., Ponce, R., (2021) O vírus da Gripe, Rev. Ciência Elem., V9(2):038
DOI http://doi.org/10.24927/rce2021.038
Palavras-chave gripe, influenza, vírus, genética, genómica, evolução, microbiologia
Resumo
A gripe é uma doença respiratória aguda, geralmente benigna, mas que pode no entanto ter grande impacto em termos de saúde pública, tanto pela sua morbilidade e mortalidade, como a nível da economia da Saúde1. Esta doença é causada por vírus da família Orthomyxoviridae que circulam nas populações humanas e animais. A gripe é uma doença sazonal que ocorre mais frequentemente nas regiões do globo onde é inverno, podendo ocorrer esporadicamente durante todo o ano nas regiões tropicais2. O vírus da gripe é constituído por várias moléculas de RNA contendo informação genética para a síntese de novos vírus, protegidas por uma estrutura de natureza proteica e lipídica. Tal como todos os outros tipos de vírus, biologicamente, são considerados parasitas intracelulares obrigatórios, pois dependem da célula hospedeira em termos energéticos e fisiológicos para a produção de novos vírus descendentes.
Estrutura e organização do vírus da gripe
Existem quatro géneros distintos de vírus da gripe, sendo que, pelo menos, três destes (Alphainfluenzavirus, Betainfluenzavirus e Gammainfluenzavirus) causam gripe nos humanos, embora com frequência (e gravidades) bem distintas3.
De um modo geral, os vírus são partículas constituídas por material genético (DNA ou RNA) englobados numa cápside de natureza predominantemente proteica. Fora das células vivas estas partículas são inertes, podendo em alguns casos permanecer no ambiente por longos períodos de tempo, como acontece com os esporos e as sementes de alguns seres vivos. No entanto, quando entram nas células hospedeiras, comportam-se como parasitas intracelulares, explorando a maquinaria celular para a síntese de novas partículas virais - a progénie viral (descendência).
Os membros da família Orthomyxoviridae possuem nucleocápsides de simetria helicoidal e genomas de RNA de cadeia simples e polaridade negativa, ou seja, de cadeia não codificante. O genoma viral apresenta-se subdividido entre seis e oito segmentos, cada um deles codificando para (uma ou duas) diferentes proteínas virais (FIGURA 1).
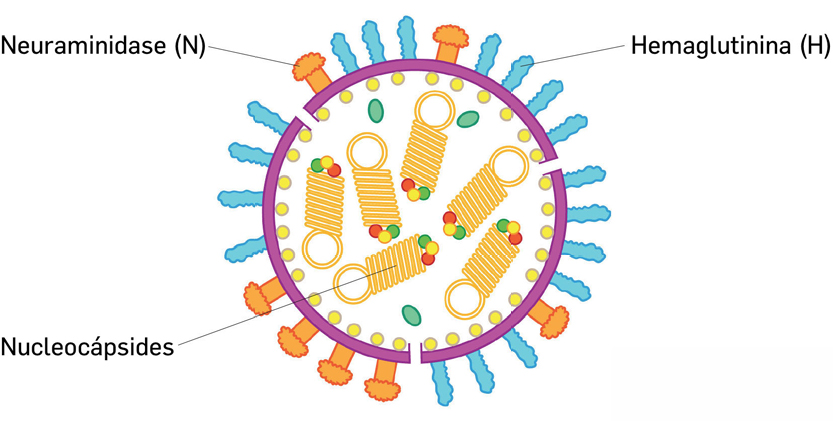
Cada segmento de RNA encontra-se compactado conjuntamente com proteínas virais formando assim nucleocápsides estruturalmente independentes (em número de oito no caso da gripe A). Uma destas proteínas virais é a enzima RNA-polimerase RNA-dependente, essencial para o ciclo replicativo do vírus pois permite a síntese de novas moléculas de RNA a partir do genoma viral. Esta função é exclusiva dos genomas de vírus de RNA.
As partículas virais são pleomórficas, i.e. não apresentam uma estrutura fixa, apesar de poderem ser esféricas, com um diâmetro de 100 nm, ou filamentosas, de 80 a 120 nm. Encontram- se ainda revestidas por um invólucro viral de natureza lipídica originado a partir da célula hospedeira infetada e que exibe na sua face externa duas glicoproteínas de origem viral: a hemaglutinina e a neuraminidase. Estas formam estruturas que se encontram projetadas cerca de 10 nm à superfície da partícula viral e possuem um papel relevante no ciclo replicativo e na patogenia viral. Funcionam como importantes antigénios de superfície, responsáveis pela variação antigénica viral e imunidade do hospedeiro.
Existem, pelo menos, três tipos de vírus da gripe com capacidade para causar doença em humanos: vírus da gripe do tipo A, B e C. Geralmente, ouvimos falar dos tipos A e B por serem os mais comuns e epidémicos4. O tipo B pode apresentar alguma variação antigénica, estando na origem de surtos epidémicos sazonais, enquanto o tipo C é antigenicamente estável e somente causa doença moderada em indivíduos imunocomprometidos.
Os vírus da gripe do tipo A, em particular, podem causar doença moderada a grave e podem também ser transmitidos a outros animais. São também antigenicamente muito variáveis e são os agentes etiológicos da maior parte das gripes e, sempre, das pandemias esporádicas. Estão subdivididos em diferentes subtipos, como por exemplo: gripe A H1N1, H3N2, H5N1 ou H7N9. Os vários subtipos são caracterizados pelas diferentes moléculas de hemaglutinina (H) e neuraminidase (N) que exibem à superfície da partícula viral.
A hemaglutinina4 é uma glicoproteína que se encontra ancorada no invólucro viral. A hemaglutinina é responsável por reconhecer especificamente e estabelecer ligação ao ácido siálico - um receptor de superfície das membranas das células epiteliais do sistema respiratório ou do trato digestivo (embora mais raramente, alguns vírus da gripe também induzam patologia gastrointestinal). A hemaglutinina tem pois como principal função ligar o vírus ao receptor da célula hospedeira. O seu nome provém dos primeiros testes de identificação dos vírus da gripe, pois estes aglutinavam as hemácias usadas no teste. As diferentes variantes são numeradas de H1 até H15, sendo que nem todas são específicas de células humanas5.
A neuraminidase4 é outra proteína que é exibida na face externa do invólucro viral, que também reconhece o ácido siálico da célula hospedeira, degradando-o e desempenhando um papel muito importante na libertação das novas partículas virais durante o ciclo de replicação viral. As diferentes variantes são igualmente numeradas, de N1 até N95.
Ciclo de replicação viral
Os vírus são partículas de dimensões sub-celulares (de dimensões inferiores à das células). Estas partículas virais, ou viriões, conseguem entrar na célula hospedeira, iniciando o ciclo de replicação viral, que consiste na síntese de todas as moléculas necessárias para a geração dos novos viriões descendentes. Pode-se dizer assim que os vírus são parasitas intracelulares obrigatórios.
O ciclo de replicação dos vírus da gripe inicia-se com o reconhecimento (adsorção) entre a célula hospedeira e o virião (FIGURA 2). Este reconhecimento é mediado pelas espículas de hemaglutinina que se encontram projetadas à superfície da partícula viral, ancoradas no invólucro viral de origem lipídica, e que se ligam aos recetores de ácido siálico na superfície celular. Havendo este reconhecimento específico, devido à sua origem lipídica, o vírus é internalizado e as nucleocápsides (contendo as moléculas de RNA) são libertadas no citosol.
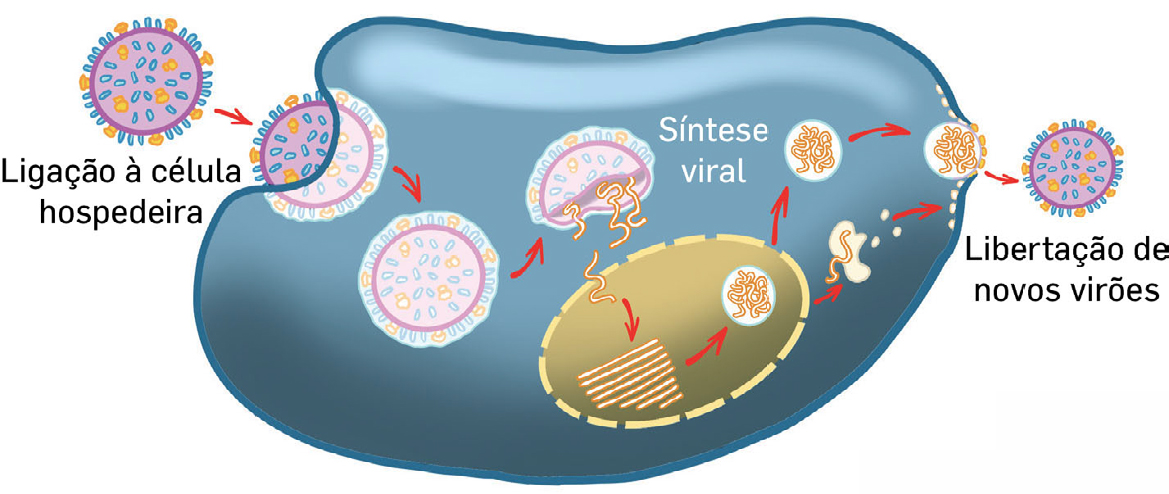
Segundo o dogma central da Biologia Molecular, a informação genética flui do DNA para o RNA e deste para as proteínas através dos mecanismos moleculares de transcrição e tradução. Há porém exceções a esta direção do fluxo da informação genética, como acontece com alguns vírus. Uma vez que os vírus da gripe são vírus de RNA de polaridade negativa, ou seja de cadeia não codificante, o genoma viral apenas serve de molde para a síntese da cadeia complementar codificante. A transcrição em RNA mensageiro viral para servir de molde para a tradução em proteínas virais, ocorre no núcleo, por ação da enzima RNA-polimerase RNA-dependente que o virião traz consigo, em associação com as nucleocápsides3, 5.
Para que haja replicação e síntese de novas cadeias de RNA de polaridade negativa para serem encapsidadas e se formarem novos viriões, é ainda necessário que seja sintetizado um intermediário replicativo de polaridade positiva, para cada um dos segmentos do genoma. Esta etapa está sujeita a uma frequência relativamente elevada de mutação génica.
O vírus da gripe é um bom modelo para o estudo e ensino dos mecanismos evolutivos. A “matéria prima” da Evolução é a variabilidade genética. O vírus da gripe evolui rapidamente e apresenta uma grande diversidade genética. Ao longo de sucessivas gerações, os vírus da gripe diversificam o seu genoma através de dois mecanismos diferentes: a deriva antigénica e a mudança antigénica por redistribuição dos segmentos do genoma. As novas variantes podem conferir ao vírus a capacidade de escapar à ação do sistema imunitário, adquirindo deste modo uma vantagem adaptativa. Assim, estas novas variantes poderão persistir e disseminar-se em populações de hospedeiros suscetíveis.
Evolução do vírus da gripe (Ecologia)
Deriva antigénica
A deriva antigénica refere-se às mutações pontuais que ocorrem e que se vão acumulando nos genes que codificam as proteínas virais, e em particular as de superfície destes vírus - aquelas que são reconhecidas pelos anticorpos.
Estas mutações ocorrem durante a replicação do material genético do vírus. De cada vez que o material genético do vírus é replicado, pela ação da RNA-polimerase RNA-dependente viral, podem ocorrer erros na síntese, originando assim mutações. Enquanto a DNA-polimerase, a enzima que sintetiza DNA, tem capacidade de proof-reading (deteção de erros durante a síntese e correção), a RNA-polimerase RNA-dependente viral não tem ação de proof-reading. Por isso, durante a replicação do material genético do vírus da gripe ocorrem muito mais erros do que, por exemplo, durante a replicação do nosso material genético.
As mutações são aleatórias e podem ocorrer em qualquer local do genoma. As consequências da mutação também podem variar - podem ter um efeito negativo (se a proteína perder função, por exemplo), neutro (se não afetar a proteína, como no caso das mutações sinónimas) ou positivo (se conferir alguma vantagem). No caso destas mutações ocorrerem nos genes que codificam as proteínas de superfície, podem permitir que o vírus escape à imunidade do hospedeiro.
Variação (mudança) antigénica e reorganização do genoma do vírus da gripe
Um mecanismo molecular que contribui para a rápida evolução do vírus da gripe é a reorganização do genoma, por redistribuição de segmentos (FIGURA 3). O genoma do vírus da gripe A encontra-se dividido em oito segmentos, cada um deles codificando, em geral, para uma ou duas proteínas/funções virais. Se uma célula suscetível de um hospedeiro estiver exposta e for infetada simultaneamente com duas, ou mais, diferentes variantes do vírus da gripe, vários segmentos de genoma poderão estar a ser replicados em simultâneo. Durante este processo diferentes nucleocápsides serão formadas em simultâneo, provenientes de estirpes virais geneticamente distintas. No entanto, aquando da libertação dos novos viriões, várias diferentes combinações de nucleocápsides poderão ser encapsidadas em cada virião. Cada virião será formado pelas oito diferentes nucleocápsides que constituem o genoma viral completo, embora, cada uma delas possa ter uma origem diferente, caso tenha havido reorganização do genoma.
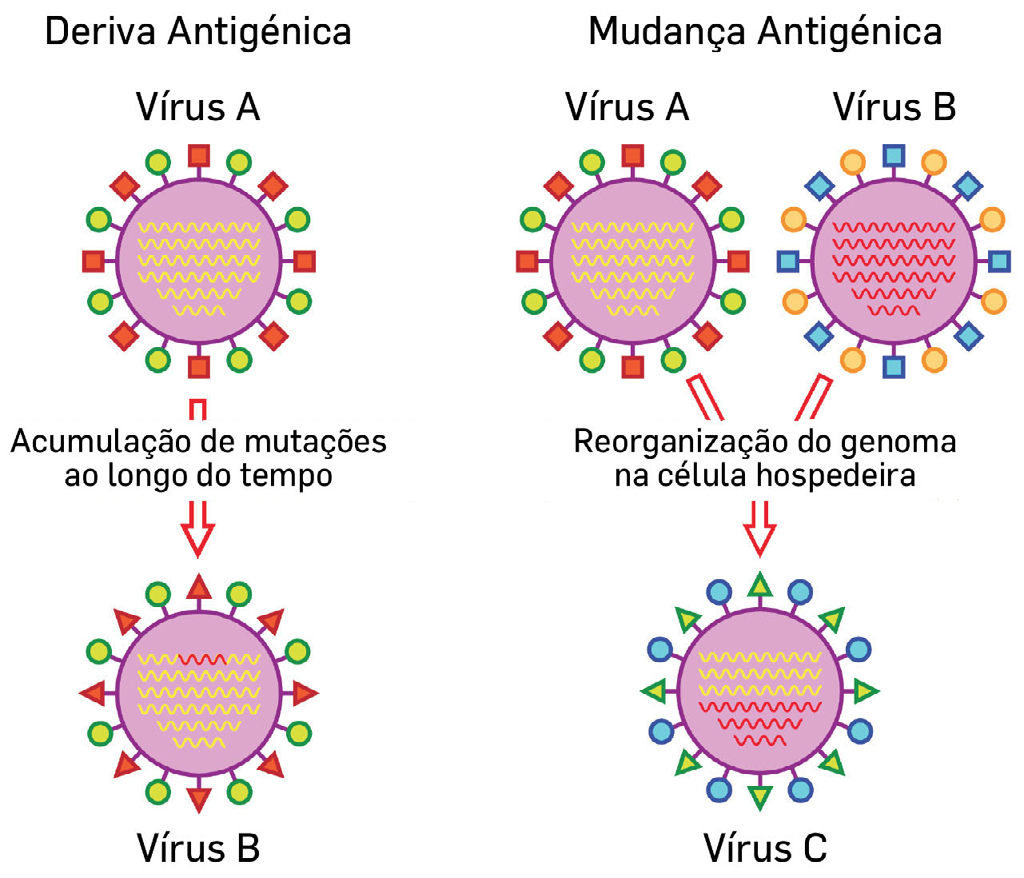
Este mecanismo de reorganização do genoma surge em resultado de alguns animais poderem ser infetados com diferentes variantes do vírus da gripe. Os suínos, por exemplo, são suscetíveis a variantes virais humanas e aviárias. Deste modo, quando as células epiteliais do trato respiratório dos porcos são infetadas simultaneamente por variantes diferentes do vírus, podem formar-se novos viriões contendo uma combinação de material genético com origem simultaneamente em vírus de gripe aviária, humana e suína.
Este rearranjo de segmentos de RNA genómico entre diferentes variantes do vírus da gripe pode levar à criação de novas combinações de proteínas H e N. Se as novas proteínas virais forem suficientemente diferentes das que se encontravam a circular entre os humanos, e para as quais ainda não exista imunidade, esta nova variante poderá, como resultado, disseminar-se rapidamente numa população humana suscetível à nova forma variante. Além disso, o rearranjo também pode resultar numa variante mais virulenta. Estas reorganizações dos genomas virais estiveram na origem das pandemias da gripe espanhola, asiática, da gripe de Hong Kong e da gripe de 20096.
Notas
As autoras agradecem ao Doutor João Piedade (Professor Auxiliar de Virologia Médica do Instituto de Higiene e Medicina Tropical) a revisão científica deste artigo.
Referências
- 1 NOGUEIRA, T. & PONCE, R., Vírus da gripe: desafios do sistema imunitário e da medicina à luz da evolução. 2019.
- 2 YANG, W. et al., Forecasting Influenza Epidemics in Hong Kong, PLoS Comput Biol, 11, e1004383. 2015.
- 3 AL-KOBAISI, M. F. et al., Sultan Qaboos Univ Med J., 7, 273–275. 2007.
- 4 Influenza A Subtypes and the Species Affected, Seasonal Influeza. 2018.
- 5 TSCHERNE, D. M. & GARCÍA-SASTRE, A., Virulence determinants of pandemic influenza viruses, J Clin Invest, 121, 6–13. 2011.
- 6 Past Pandemics, Pandemic Influenza. 2019.
Este artigo já foi visualizado 23341 vezes.



