Estudo das proteases Cat B e L
Proteínas humanas fundamentais para a replicação do vírus SARS-CoV-2, causador da pandemia COVID-19
📧 , 📧 , 📧
- * Universidade do Porto
- ɫ Universidade do Porto
- ‡ Universidade do Porto
Referência Cunha, M., Fernandes, P. A., Ramos, M. J., (2022) Estudo das proteases Cat B e L, Rev. Ciência Elem., V10(1):008
DOI http://doi.org/10.24927/rce2022.008
Palavras-chave Proteínas, aminoácidos, enzimas, centro ativo, vírus, SARS-Cov-2
Resumo
A entrada do vírus SARS-CoV-2 no corpo humano necessita da ação de proteases de cisteína — catepsinas B e L. Estas estão envolvidas na clivagem da proteína espicular do vírus, passo essencial para que o vírus se instale, desenvolva e multiplique na célula hospedeira. Pensa-se que a inibição das catepsinas B e L possa ser uma nova forma de diminuir a propagação desta doença na população mundial.
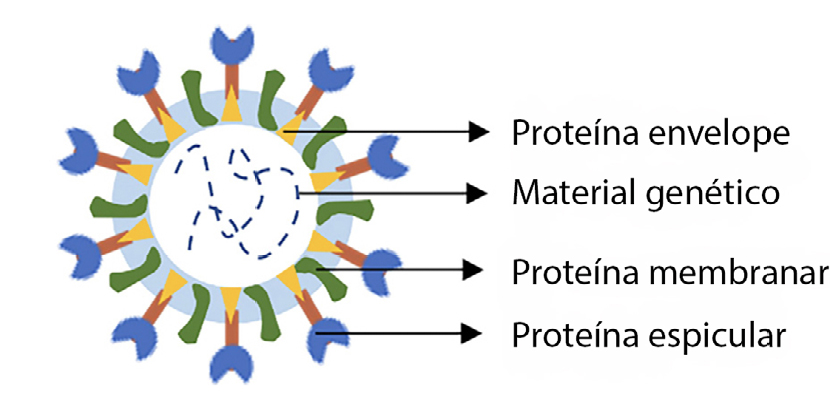
Identificada pela primeira vez na cidade de Wuhan, na China, em 2019, a doença vírica causada pelo SARS-CoV-2 proliferou pelo mundo, tendo sido denominada de COVID-19. Esta doença pode ser traduzida por ligeiros sintomas, sendo esta a sintomatologia mais comum em casos confirmados. Porém, muitos outros experimentam sintomas muito fortes e em casos extremos, morte. O vírus SARS-CoV-2 é o causador da síndrome respiratória aguda grave, propaga-se através de gotículas projetadas pelo nariz ou pela boca, quando tossimos, espirramos, falamos ou até mesmo quando respiramos. A sua taxa de transmissão é elevada, o que faz dele uma verdadeira ameaça à população, e o seu combate ou prevenção de o contrairmos passa pelo uso de máscara, distância social, lavagem das mãos com sabão várias vezes ao dia, ou, em alternativa, a desinfeção das mãos com produtos à base de álcool. O SARS-CoV-2 é conhecido pela sua forma esférica e é constituído por proteínas envelope (E), proteínas membranares (M), proteínas espiculares (S, do inglês spike) e pelo material genético (FIGURA 1). As proteínas M e S têm um importante envolvimento na instalação e replicação do vírus no hospedeiro.
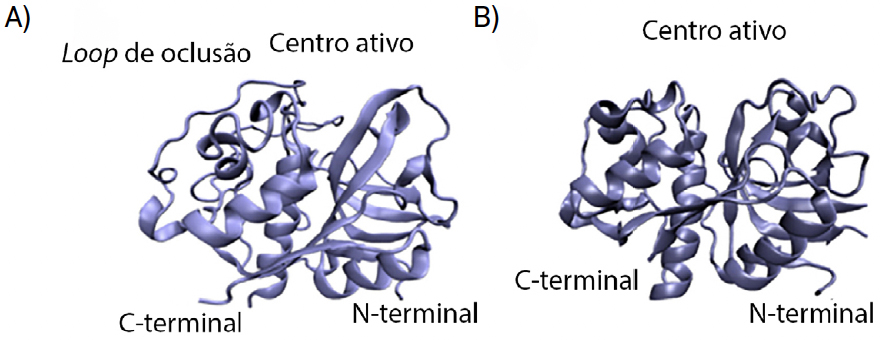
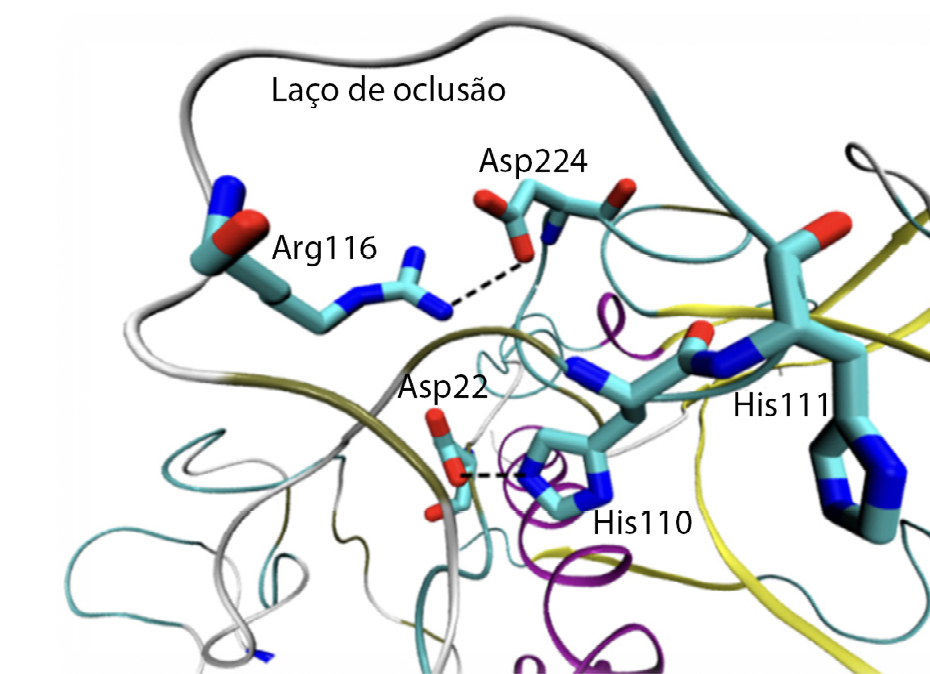
As catepsinas são enzimas, ou seja, são moléculas que aceleram reações químicas. Tratam-se de enzimas do tipo “protease de cisteína”, visto que contêm um resíduo de cisteína reativo no seu centro ativo, e encontram-se na maioria das células humanas. Estas enzimas têm um papel importante na degradação intracelular de proteínas. Para a sua atividade ser maximizada, o pH deverá ser acídico, entre 4,5 e 5,04. A catepsina L é, geralmente, encontrada em todos os tipos de células e é composta por 220 resíduos de aminoácido e tem como função primordial a degradação de proteínas. A catepsina B apresenta estrutura e função semelhante à catepsina L; porém, apresenta um segmento adicional peptídico, denominado laço de oclusão (do inglês, occlusion loop). Este segmento é constituído por 20 resíduos de aminoácido e tem como função o bloqueio do centro ativo da catepsina (FIGURA 2), devido ao posicionamento estratégico de duas histidinas (His110 e His111). Também os resíduos His110 e Asp22 criam entre si uma ponte salina que confere uma certa rigidez ao laço, sendo esta ligação a responsável pela influência do pH na atividade da catepsina. Os resíduos Arg116 e Asp224 estabelecem também entre si interações, assim como os resíduos de cisteína 108 e 109, que formam uma ponte dissulfureto.
O laço de oclusão pode ser encontrado de duas formas: aberto e fechado. Quando o laço se encontra na posição aberta (antes da maturação da catepsina B) as cadeias laterais dos resíduos His110 e Asp224, bem como as cadeias laterais de His111 e Asp224, encontram-se muito próximas entre si. Após a maturação, estas interações são removidas e são construídos novos pontos de ligação. A FIGURA 3 ilustra interações que existem entre os resíduos quando o laço se encontra na posição fechada.
Ambas as catepsinas apresentam centros ativos contendo cisteína e histidina, porém o tamanho e forma destes locais de ligação apresentam diferenças, ainda que ligeiras.
A replicação do vírus divide-se em três passos fundamentais. Na primeira etapa dá-se a entrada do vírus na célula, de seguida dá-se a replicação do RNA do vírus e, por último, a montagem viral ocorre e o vírus abandona a célula hospedeira. O primeiro passo para a entrada do vírus nas células hospedeiras é a ativação da sua proteína espicular. A proteína espicular é composta por duas subunidades, S1 e S2 (FIGURA 4).
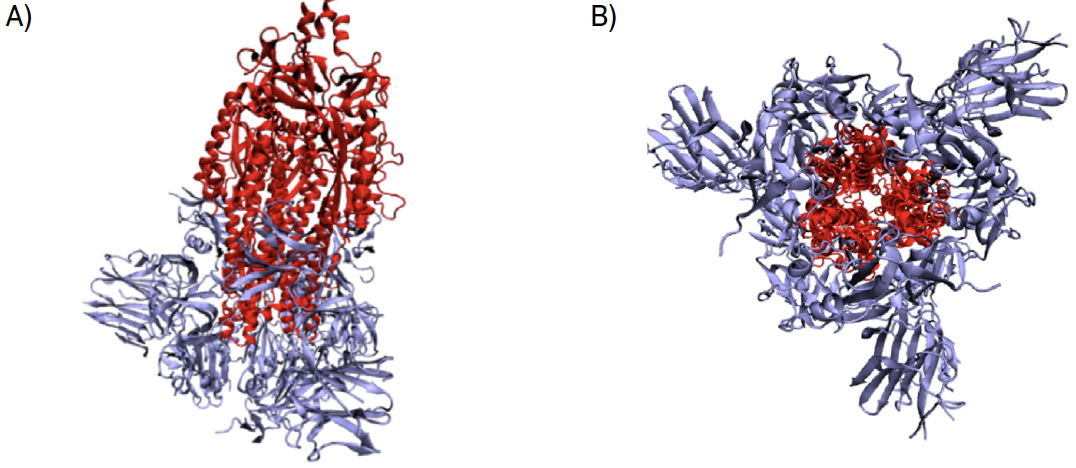
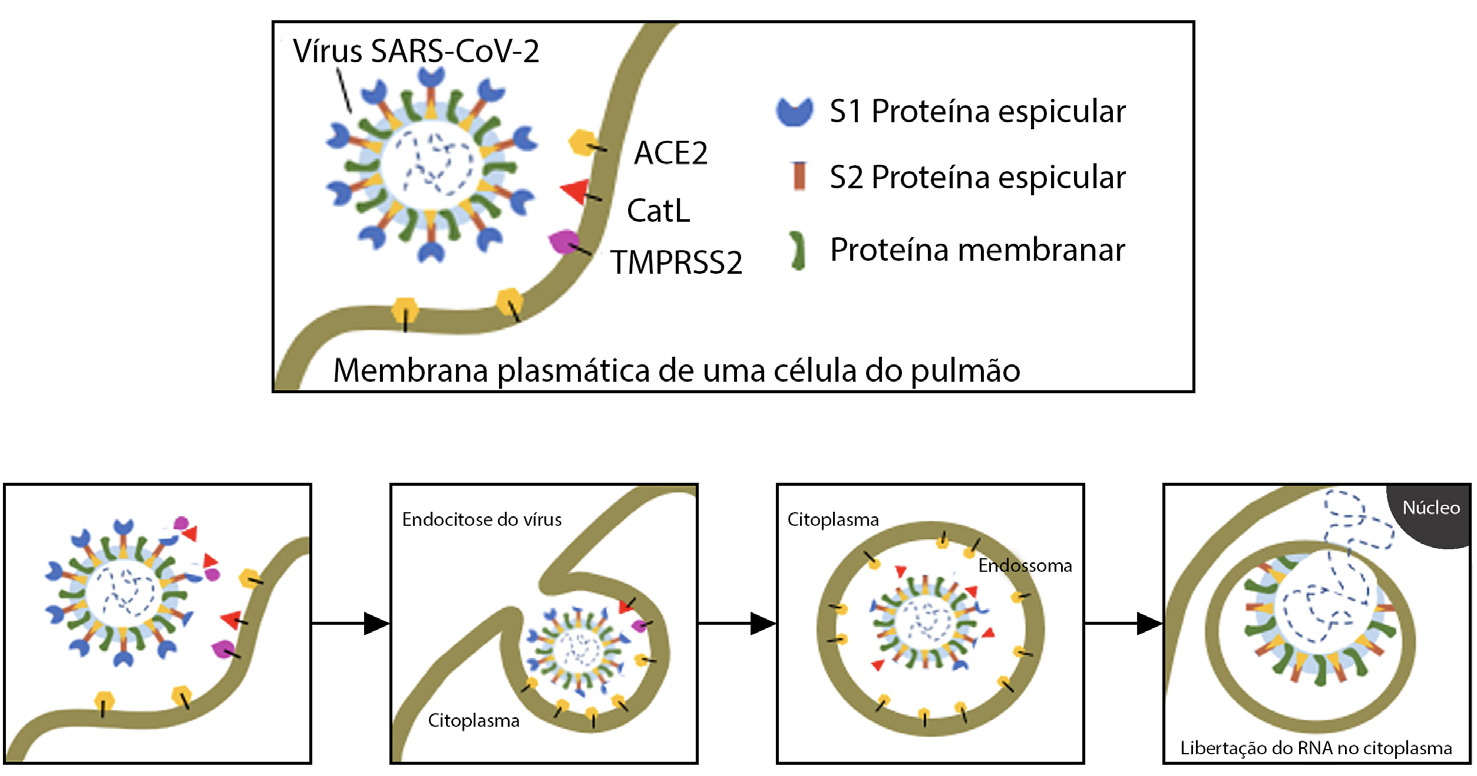
Durante a entrada do vírus na célula hospedeira, estas duas subunidades são clivadas entre os resíduos Thr696 e Met697, por diversas proteínas — pela protease transmembranar serina 2 (TMPRSS2), por pro-proteínas convertases (como a furina), por proteases extracelulares (como as elastases) mas, principalmente, pela catepsina L1. A catepsina L é a principal responsável pela clivagem da glicoproteína devido ao seu pH acídico que é muito propício para o efeito2. A entrada do vírus pode dar-se por endocitose, difusão simples ou ainda por ligação a recetores3. O processo endocítico apresenta- -se representado na FIGURA 5.
A subunidade S1 do vírus liga-se ao recetor superficial da célula hospedeira, ACE2 (do inglês angiotensin-converting enzyme 2) e, de seguida, a subunidade S2 sofre uma outra clivagem para promover a ativação do péptido de fusão hidrofóbico, fazendo com que este se funda na membrana, introduzindo o seu RNA na célula hospedeira1. Durante esta fusão na membrana, continua a degradação da subunidade S1 pela enzima Cat L. Relativamente ao papel da catepsina B, ainda muito pouco se sabe.
Visto que as catepsinas têm um papel fulcral na entrada do vírus, pensa-se que a sua inibição seja uma potencial forma de travar a propagação do mesmo, em especial a catepsina L. A inibição das catepsinas B e L é feita através de inibidores enzimáticos. Estes são substâncias que reduzem a velocidade de reações catalisadas por enzimas. Em condições normais, a enzima forma um complexo com o substrato — complexo enzima-substrato — catalisando, pois, reações que têm lugar no centro ativo da enzima. O inibidor formará uma ligação com a enzima mais forte e mais estável do que a ligação da enzima ao substrato, fazendo então com que a sua função cesse.
Os mecanismos de inibição podem ser reversíveis, compostos maioritariamente por interações não-covalentes, ou irreversíveis, compostos por interações covalentes.
É importante frisar que, segundo as regras de Lipinsky, é aconselhável que um inibidor seja uma molécula pequena, com uma massa molecular menor que 500 g mol-1. Os inibidores não devem contar com mais de 5 átomos dadores de pontes de hidrogénio na sua estrutura (ligações N-H e O-H) nem com mais de 10 átomos aceitadores de pontes de hidrogénio (átomos de azoto e de oxigénio). Se estas regras não forem observadas, é pouco provável que os inibidores possam ser tomados oralmente, em comprimidos, e cheguem às células-alvo em quantidade significativa.
Posto isto, seguem-se alguns compostos químicos que poderão funcionar como potenciais inibidores às catepsinas B e L.
O composto E-64 ou, nome IUPAC, 1-[n-[(L-3-trans-carboxioxirano-2-carbonil)-L-leucil] amino]-4-guanidinobutano, um inibidor irreversível de proteases de cisteína, liga-se especifica e covalentemente à catepsina B. Este inibidor contém na sua estrutura um epóxido (evidenciado a preto, na FIGURA 6) que, no centro ativo da catepsina, estabelece uma interação covalente com um grupo tiol da cisteína 29.
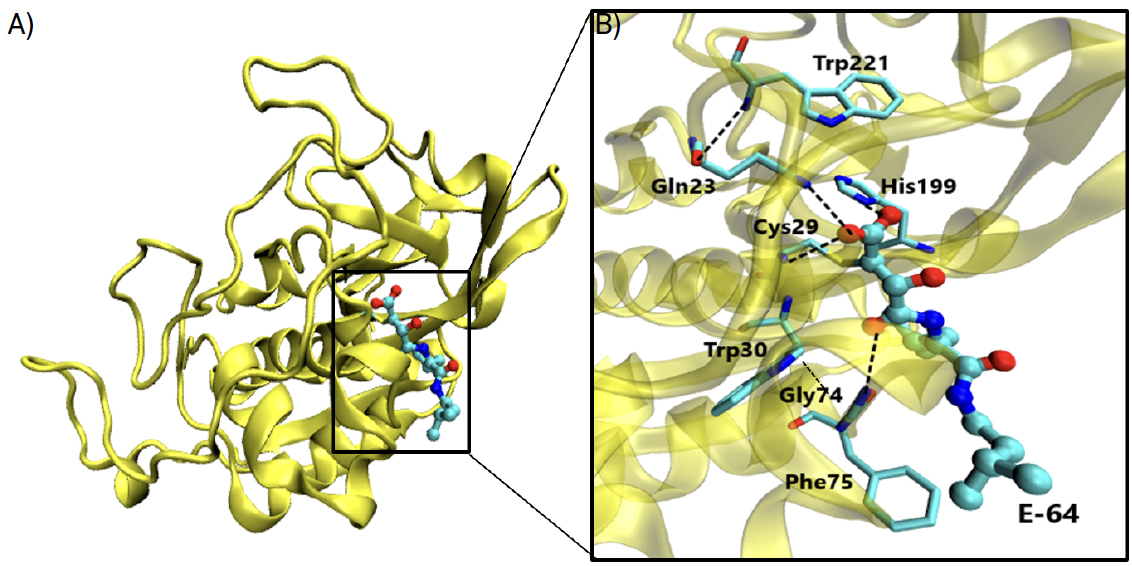
A especificidade deste inibidor é estudada com base nas interações do inibidor com o centro ativo, e, por esse motivo, já foram desenvolvidos derivados do E-64 para catepsinas específicas. No entanto, o composto E-64 não costuma ser utilizado a nível clínico devido à substituição por compostos mais específicos – por exemplo, para a catepsina B, utiliza- -se o composto CA0744. A FIGURA 6 consiste numa visão global, FIGURA 6A), e detalhada, FIGURA 6B), do centro ativo da catepsina B e das interações que o inibidor E-64 nele pode estabelecer. Na mesma figura estão evidenciadas várias ligações da molécula E-64 a resíduos do centro ativo da catepsina B, tais como ligações hidrogénio (que são ligações intermoleculares que contam com alguma intensidade, e, portanto, é necessária mais energia para a sua quebra) entre os oxigénios da E-64 e os átomos de azoto dos resíduos Gly74, Gln23 e Gly29.
É também importante evidenciar as interações que existem na cavidade oxianiónica, (do inglês oxyanion hole), que são compostas pelo oxigénio 17 da molécula do E-64 e pelos átomos de azoto dos resíduos Cys29 e Gln23, todas elas muito importantes para que a reação catalítica se dê. Existem também ligações que contribuem de forma indireta para a estabilização da cavidade oxianiónica, estando entre elas o átomo de azoto da Trp221 e o oxigénio da Gln23. Os pares iónicos do inibidor e da enzima podem igualmente formar interações hidrofóbicas e eletrostáticas, protagonizadas principalmente por átomos de carbono do inibidor com os resíduos Gly74 e Phe755.
O composto CA074, 4 [N-(L-3-trans-propilcarbamoiloxirano-2-carbonil)-L-isoleucil-L- prolina], apresentado na FIGURA 7, é um inibidor específico para a catepsina B, derivado de E-64 que possui um epóxido, tal como E-64, ligando-se covalentemente à cisteína 29, e faz parte do centro ativo da catepsina B.
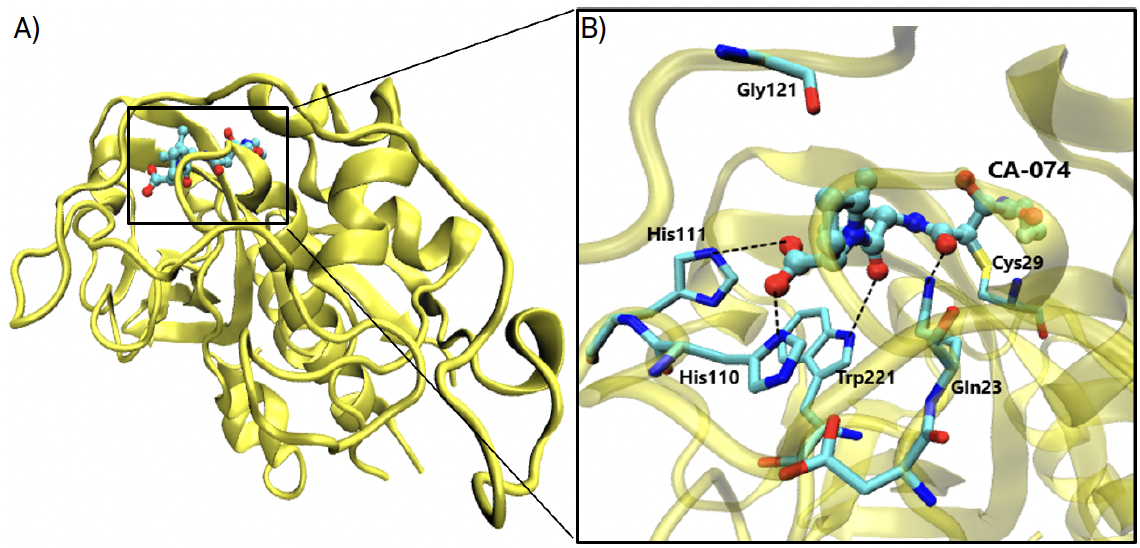
Foi testada a capacidade inibitória do CA-074 sem o grupo epóxido, tendo-se obtido uma perda significativa de atividade inibitória e concluído que a ligação covalente entre o epóxido e Cys29 é absolutamente crucial para que a inibição se dê com eficiência, sem menosprezar outras interações auxiliares6. A cavidade oxianiónica da estrutura CA-074 complexada com a catepsina B é constituída pelo grupo carbonilo do inibidor CA-074 e pelos átomos de azoto dos resíduos Cys29 e Gln23, cujas interações hidrofóbicas são necessárias para que o inibidor cumpra a sua função. As ligações de hidrogénio evidenciadas na FIGURA 7 existem entre os grupos imidazol dos resíduos His110 e His111 (que constituem o laço de oclusão) e entre os dois oxigénios do grupo carboxílico do inibidor7, 8, 9, sendo extremamente importantes para a ligação específica que caracteriza este inibidor à catepsina B. Este inibidor já foi e continua a ser alvo de grandes apostas contra o cancro da mama (devido ao envolvimento das catepsinas B e L nesta doença), embora estudos para o tratamento da COVID-19 estejam ainda a ser averiguados.
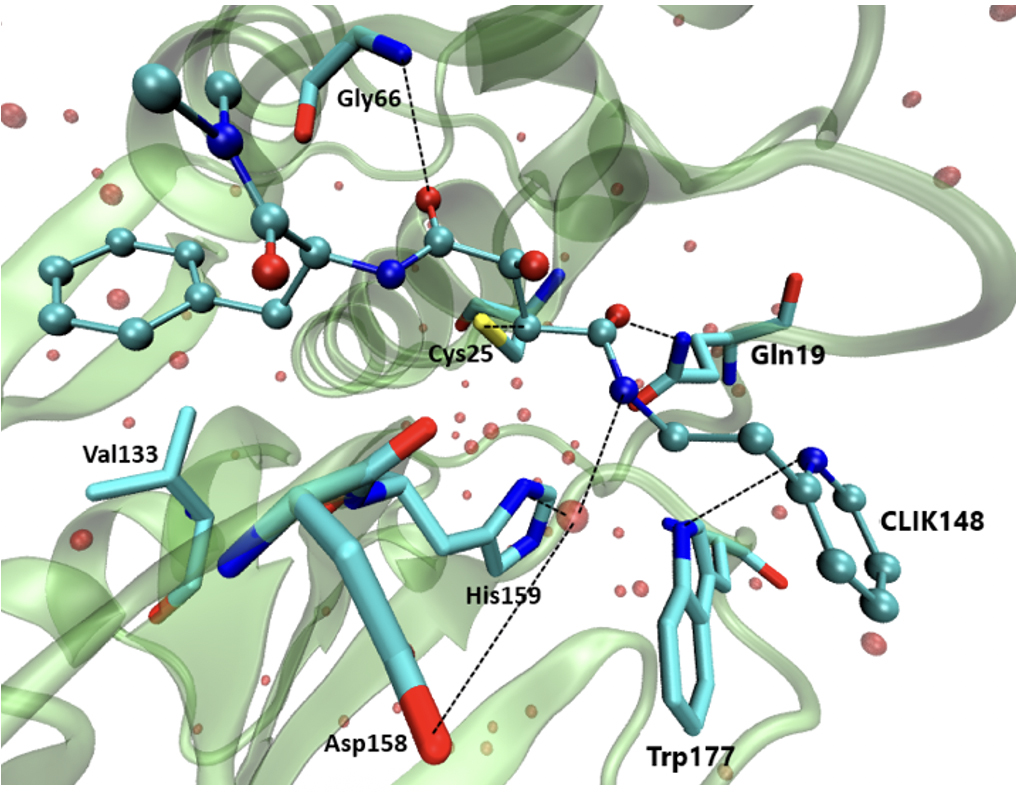
O composto CLIK148, derivado da série CLIK, com nome IUPAC piridiletilamido-N-(- transcarbamoiloxirano-2-carbonil)-L-fenilalanina-dimetilamida, é conhecido por ser um inibidor de enzimas com estrutura semelhantes à protease de cisteína papaína, como, por exemplo, a catepsina L. Pertence ao grupo dos epoxissucinato e a inibição tem lugar devido a uma reação de alquilação do átomo de enxofre pertencente a um resíduo de cisteína do centro ativo das proteases de cisteína. Isto provoca a abertura do epóxido do inibidor, de modo a ocorrer um ataque nucleofílico por parte de um dos carbonos do grupo epóxido, formando uma ligação tioéster. Esta interação é covalente e a reação é irreversível.
Para um estudo com maior aproveitamento deste potencial inibidor contra a catepsina L, é necessária avaliar a semelhança entre a papaína e a catepsina L. A papaína é também uma protease de cisteína e hidrolisa ligações amida através de uma tríade catalítica, geralmente formada por resíduos de cisteína, histidina e asparagina, sendo este último muitas vezes substituído por um outro aminoácido, o aspartato. A cavidade oxaniónica situa-se no centro da estrutura da papaína, bem como nas catepsinas B e L, onde se forma uma fenda provocada pela separação de dois domínios. A partir da estrutura do inibidor CLIK148 complexado com a papaína, é possível prever o mecanismo e as ligações que poderão existir na inibição da catepsina L, visto que a estrutura, centros ativos e disponibilidade da papaína e da catepsina são muito semelhantes. A partir de estudos publicados, sabemos que este inibidor apresenta inibição específica à catepsina L (inibição a 60%, utilizando uma concentração de apenas 10-7 M de inibidor), mas o contrário foi verificado relativamente a outras catepsinas, nas quais a inibição não foi observada. É importante estudar estas interações com a papaína para que mais estudos sejam feitos para inibidores específicos da catepsina L10. Entre as interações existentes, destaca-se a interação aromática entre o anel piridina do inibidor e o resíduo Trp177. Ligações de hidrogénio estão presentes entre um átomo de azoto do resíduo Gln19 e um átomo de oxigénio do inibidor e de modo semelhante, um átomo de azoto do resíduo Gly66 possui também uma interação com um átomo de oxigénio do CLIK148. Existe uma molécula de água próxima do resíduo His159, que estabelece também ligações de hidrogénio, melhorando assim o índice de inibição. Além da ligação com His198, esta molécula de água forma duas outras interações, uma com o resíduo Asp159 e outra com o inibidor. Por último, o resíduo Cys25 forma uma ligação covalente, através do seu átomo de enxofre, com um átomo de carbono do inibidor. Apesar de ser potencialmente crucial, para o tratamento da COVID-19, a inibição da catepsina L, bem como para outras doenças onde a catepsina L atue, é fulcral o estudo da papaína com o inibidor CLIK148 para que se continuem a desenvolver inibidores específicos para esta catepsina, visto que estas interações não estão ainda suficientemente claras. Pensa-se que a especificidade à catepsina L se deve a uma interação hidrofóbica do anel piridina do CLIK148 com o grupo N-terminal da catepsina L. Esta afirmação é feita devido a evidências de que CLIK148 não inibe a catepsina B, porque o seu laço de oclusão bloqueia a interação do anel de pirimidina com o N-terminal.
Já foi ponderado considerar a cloroquina e seus derivados como possíveis fármacos para o tratamento da COVID-19. Estes compostos são muito utilizados nos tratamentos da malária e doenças reumáticas. Os mecanismos de ação da cloroquina e hidrocloroquina passariam pela inibição da endocitose, bloqueando a maturação dos endossomas. Estes, por sua vez, iriam desencadear a fusão da membrana celular3. Porém, a nível clínico, ainda não foi descoberta atividade viral relevante contra o SARS-CoV-2 fazendo, assim, com que não seja aconselhável o uso destas substâncias no tratamento da COVID-19.
O uso de fármacos será o próximo grande passo no estudo deste vírus, uma vez que as vacinas já se encontram a ser distribuídas à população mundial e a inibição de proteases de cisteína, tem vindo a revelar-se um grande ponto de partida para esse fim.
Ainda não foram conseguidos inibidores para a catepsina B com hipóteses de serem testados e usados clinicamente, devido à pouca biodisponibilidade que apresentam, efeitos secundários inesperados e indesejados ou até mesmo, alta toxicidade11. Contudo, há possibilidade de que o inibidor específico para catepsina B já apresentado neste trabalho, o CA-074, possa constituir eventualmente, com algumas alterações na sua estrutura, um fármaco. O papel da catepsina B, no ser humano infetado, bem e como o ciclo de vida do SARS-CoV-2 e suas inibições, continuam a ser estudados, porque ainda pouco se sabe acerca deste assunto. Pensa-se também que a catepsina L, inibida, poderá ser um grande ponto de partida contra a COVID-19. Neste caso, o CLIK148 poderá apresentar bons resultados devido ao seu grande potencial em inibir a catepsina L. Contudo, mais estudos têm de ser elaborados com estes e demais compostos, para que as suas interações com o centro ativo da enzima, bem como a sua biodisponibilidade e toxicidade para o Homem, sejam clarificados.
Referências
- 1 GOMES, C. P. et al., Cathepsin L in COVID-19: From Pharmacological Evidences to Genetics, Front Cell Infect Microbiol. 2020.
- 2 LIU, T. et al., Cathepsin L-selective inhibitors: A potentially promising treatment for COVID-19 patients, Pharmacol, 213, 107587. 2021.
- 3 JOSHI, S. et al., Tackling SARS-CoV-2: proposed targets and repurposed drugs, Future Med Chem., 12, 17, 1579–601. 2020.
- 4 WILDER, C. L. et al., Differential cathepsin responses to inhibitor-induced feedback: E-64 and cystatin C elevate active cathepsin S and suppress active cathepsin L in breast cancer cells, Int J Biochem Cell Biol., 79, 199–208. 2016.
- 5 YAMAMOTO, A. et al., Structural basis for development of cathepsin B-specific noncovalent-type inhibitor: Crystal structure of cathepsin B-E64c complex, Biochim Biophys Acta - Protein Struct Mol Enzymol, 1597, 2, 244–51. 2002.
- 6 SHENOY, R. T. & SIVARAMAN, J., Structural basis for reversible and irreversible inhibition of human cathepsin L by their respective dipeptidyl glyoxal and diazomethylketone inhibitors. 2010.
- 7 YAMAMOTO, A. et al., Substrate Specificity of Bovine Cathepsin B and Its Inhibition by CA074, Based on Crystal Structure Refinement of the Complex, J Biochem, 127. 2000.
- 8 FUJISHIMA, A. et al., The crystal structure of human cathepsin L complexed with E-64, FEBS Lett, 21, 407, 1, 47–50. 1997.
- 9 YAMAMOTO, A. et al., Binding mode of CA074, a specific irreversible inhibitor, to bovine cathepsin B as determined by X-ray crystal analysis of the complex, J Biochem., 121, 5, 974–7. 1997.
- 10 TSUGE, H. et al., Inhibition Mechanism of Cathepsin L-Specific Inhibitors Based on the Crystal Structure of Papain – CLIK148 Complex, 416, 411–6. 1999.
- 11 MIRKOVI?, B. et al., Novel Mechanism of Cathepsin B Inhibition by Antibiotic Nitroxoline and Related Compounds, ChemMedChem, 6, 8, 1351–6. 2011.
Este artigo já foi visualizado 3387 vezes.



