Identificação Taxonómica em Biologia usando Inteligência Artificial
📧 , 📧 , 📧 , 📧 , 📧 , 📧
- * DCC/ FC/ Universidade do Porto
- ɫ DCC/ FC/ Universidade do Porto
- ‡ DCC/ FC/ Universidade do Porto
- + DCC/ FC/ Universidade do Porto
- # DCC/ FC/ Universidade do Porto
- § DCC/ FC/ Universidade do Porto
Referência Lopes, L. M. B., Marques, E. R. B., Mamede, T., Filgueiras, A., Marques, M., Coutinho, M., (2022) Identificação Taxonómica em Biologia usando Inteligência Artificial, Rev. Ciência Elem., V10(4):050
DOI http://doi.org/10.24927/rce2022.050
Palavras-chave identificação taxonómica; biologia; inteligência artificial; ciência cidadã
Resumo
Neste artigo apresentamos o projeto Biolens cujo objetivo é permitir a identificação automática de espécies biológicas a partir de fotografias usando técnicas de inteligência artificial. O Biolens disponibiliza uma aplicação Web que atualmente permite a identificação de espécies de libélulas e libelinhas, borboletas diurnas e noturnas, e plantas. Adicionalmente, uma aplicação piloto iOS integra esta funcionalidade com o registo e disseminação de avistamentos de espécies de borboletas diurnas.
A ubiquidade dos telemóveis, a sua capacidade computacional e a elevada qualidade dos sensores fotográficos que os equipam abrem imensas possibilidades à participação dos cidadãos em atividades científicas, um conceito designado por Ciência Cidadã (Citizen Science). É neste contexto que surge o Biolens1, um projeto para classificação automática de imagens de espécies biológicas usando técnicas de inteligência artificial.
A aplicação Web desenvolvida pelo projeto sugere uma identificação taxonómica para um animal ou planta cuja fotografia é carregada pelo utilizador. Assim, um utilizador pode não só recolher informação relevante sobre uma espécie (por exemplo, fotografia, data, local), mas tem também a possibilidade de saber, em geral com um grau de confiança elevado, qual o nome científico do animal ou planta que acabou de registar. Atualmente, a aplicação inclui modelos de classificação para diferentes grupos taxonómicos da fauna e flora de Portugal: libélulas e libelinhas (Dragonlens), borboletas diurnas (Lepilens) e noturnas (Mothlens) e plantas (Floralens) (FIGURA 1). O trabalho foi iniciado com o desenvolvimento do Lepilens2, 3. Posteriormente, foram desenvolvidos o Mothlens, DragonLens, e, mais recentemente, o FloraLens4, 5.
Como foi referido, a identificação taxonómica das imagens recorre a ferramentas de inteligência artificial, nomeadamente de machine learning, cujo objetivo é “ensinar” conceitos aos computadores por forma a que estes possam reter esse conhecimento e aplicá-lo noutros contextos.
No caso concreto do projeto Biolens, utilizam-se redes neuronais profundas (deep learning) que tentam simular em computador a forma como o nosso cérebro aprende e sintetiza conhecimento através de redes de neurónios que processam e trocam informação entre si.
No caso do Mothlens, por exemplo, o processo de aprendizagem passa por fornecer ao computador um conjunto de imagens de mariposas com a respetiva classificação taxonómica correta (indicada ou verificada por especialistas). Este conjunto funciona como uma referência para a rede neuronal aprender as diferenças e semelhanças entre as espécies representadas. Depois de um processo de treino, tipicamente bastante intensivo em termos computacionais e que pode demorar várias horas ou mesmo dias, a configuração final da rede designada por modelo, sintetiza o conhecimento adquirido. O modelo pode então receber imagens de mariposas e responder com sugestões de identificação taxonómica (FIGURA 2).
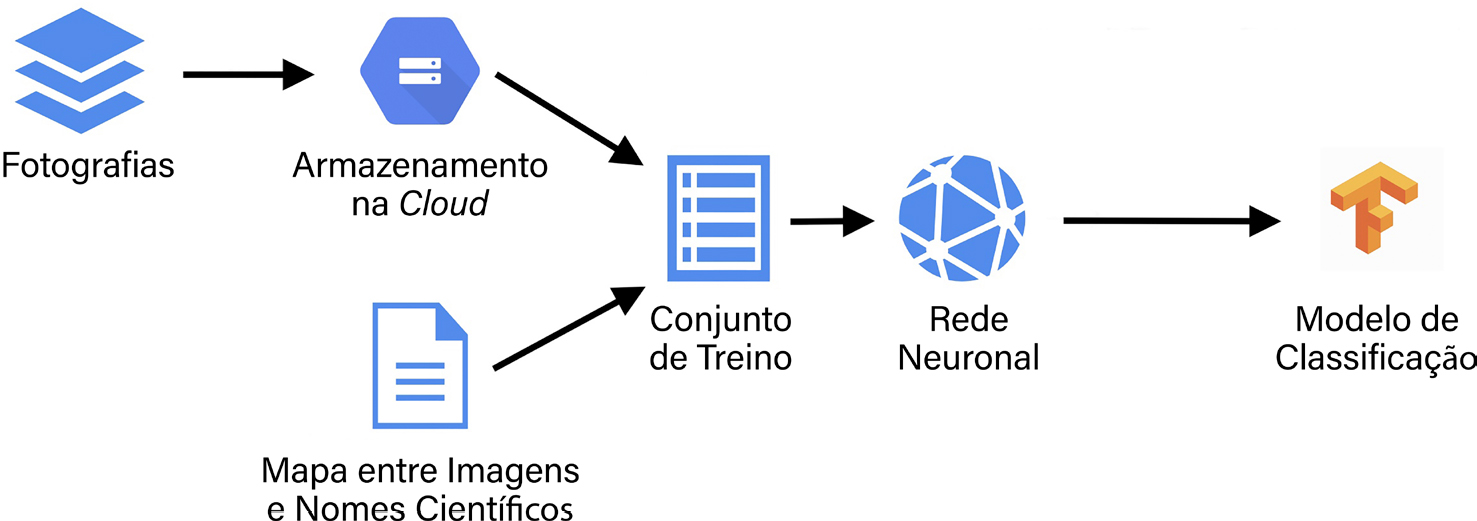
Os modelos fornecidos pelo Biolens foram treinados usando os serviços da Google Cloud, mais concretamente a plataforma Google AutoML6 e a biblioteca open-source TensorFlow para machine learning7. A arquitectura das redes neuronais derivadas pelo Google AutoML é especificamente dirigida à utilização em dispositivos com recursos computacionais limitados8, não só computadores com recursos modestos, mas também dispositivos como telemóveis ou tablets da forma discutida mais abaixo. Isto permite, por exemplo, que a aplicação Web do Biolens esteja alojada numa máquina virtual com apenas 4 GB de RAM e 2 CPUs.
As imagens utilizadas no treino das redes neuronais foram extraídas de conjuntos de dados (datasets) publicados no Global Biodiversity Information Facility (GBIF) por plataformas de ciência cidadã bem conhecidas: o iNaturalist9, o Observation.org10 e o Pl@ntNet11. Além de dados disponíveis via GBIF, para o FloraLens empregamos ainda o conjunto de imagens de excelente qualidade, identificadas por especialistas, do projeto FloraOn12 da Sociedade Portuguesa de Botânica. Para o Lepilens e Mothlens foram utilizados outros repositórios com fotografias para as quais a classificação havia sido validada por especialistas, em particular o Lusoborboletas13.
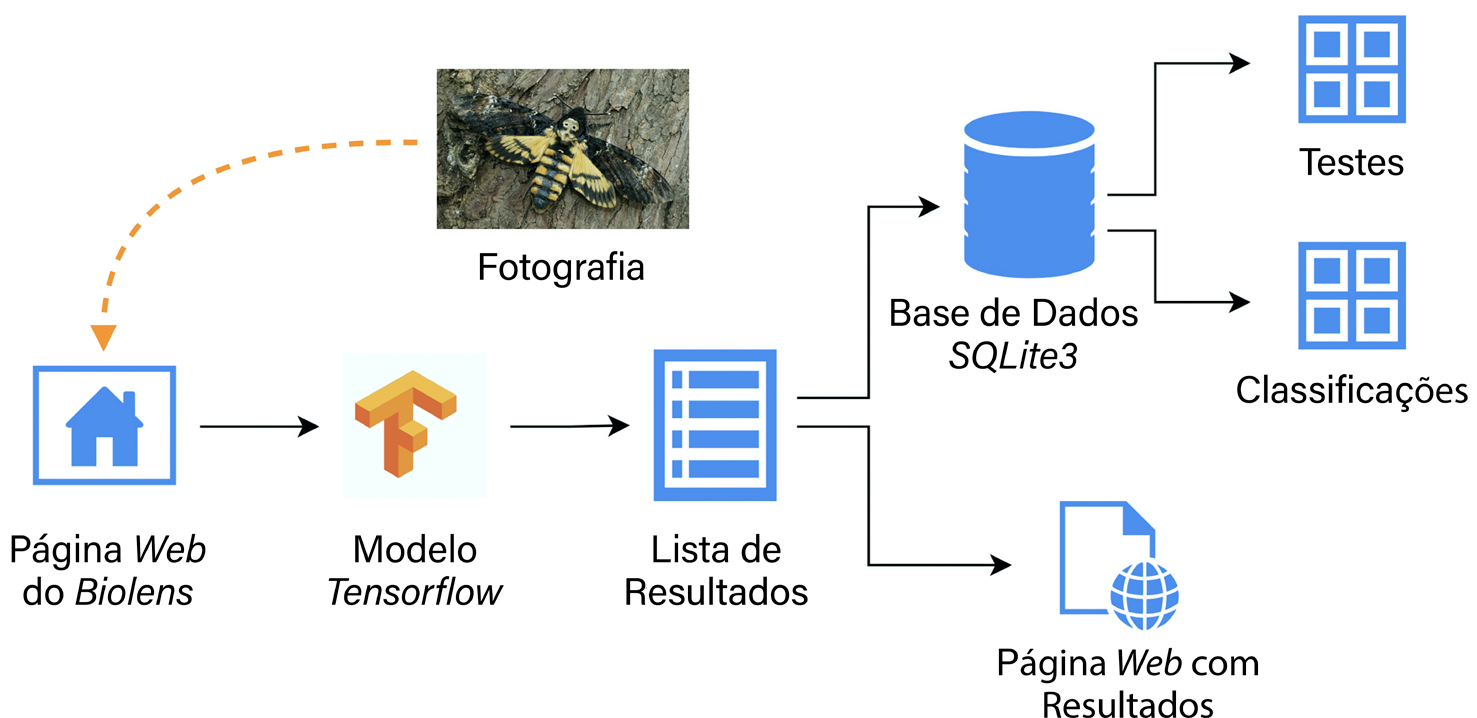
A aplicação Web do Biolens alberga os modelos supracitados para identificar de forma automática imagens de animais ou plantas submetidas por utilizadores. Para cada imagem, a aplicação selecionada gera uma lista de sugestões de identificação ordenadas por ordem decrescente de confiança. Estes resultados são apresentados aos utilizadores numa página web gerada dinamicamente e guardados numa base de dados no servidor, juntamente com a imagem e a data de submissão (FIGURA 3). Isto é possível graças ao uso dos modelos gerados pelo Google AutoML na variante TensorFlow Lite, um formato otimizado para a utilização em dispositivos com recursos computacionais limitados, não só computadores com recursos modestos, mas também dispositivos como telemóveis ou tablets da forma discutida mais abaixo.
A aplicação Web é albergada num computador servidor modesto com apenas 4 GB de RAM e 2 CPUs.
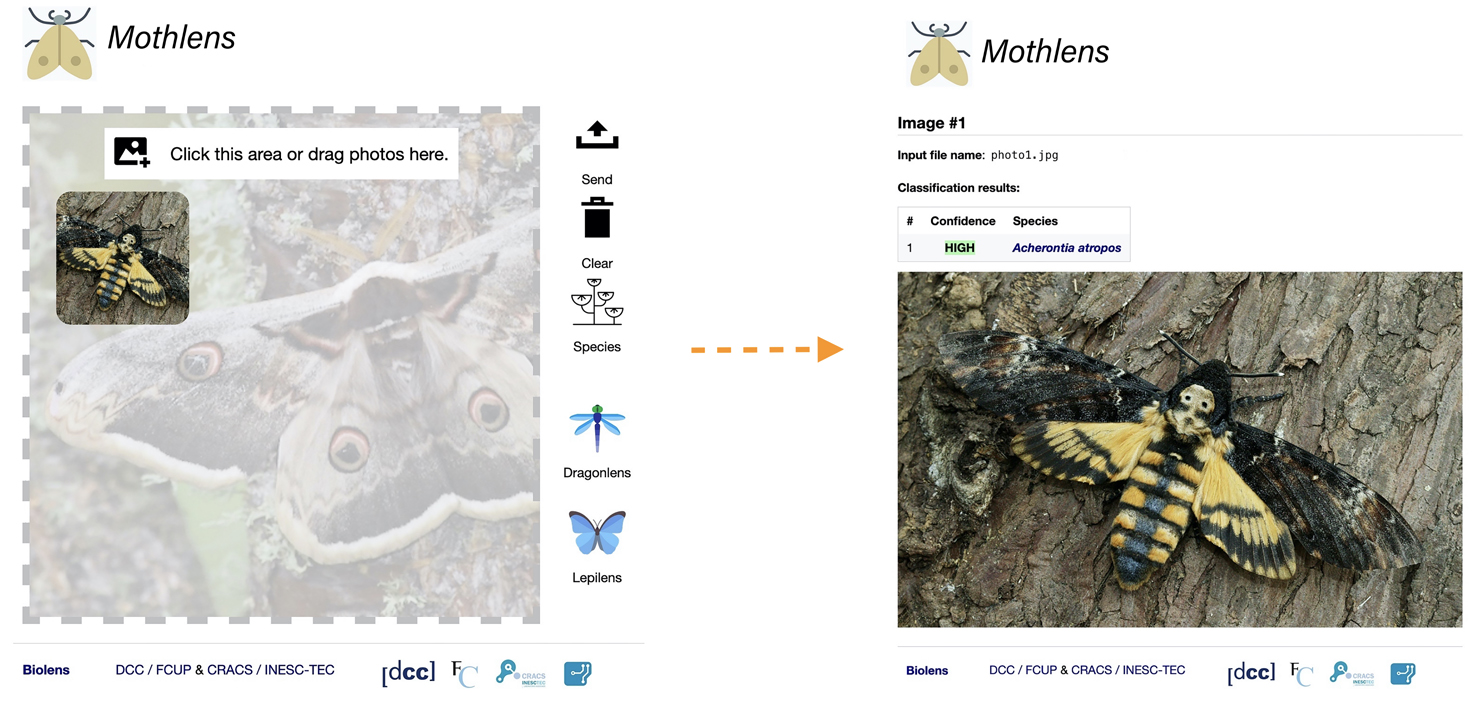
Naturalmente, os modelos são falíveis. Defeitos de construção no conjunto de treino, imagens mal identificadas no dito conjunto ou imagens de baixa qualidade submetidas pelos utilizadores finais, por exemplo, podem levar a que os modelos produzam sugestões incorretas. Os resultados devem por isso ser encarados como “sugestões informadas” e não como identificações definitivas, as quais devem sempre passar pelo crivo de um ou mais especialistas humanos (FIGURA 4).
No seguimento do desenvolvimento da aplicação Web Biolens, exploramos também a possibilidade de oferecer o Biolens através de uma aplicação móvel.
Apesar das aplicações web poderem ser utilizadas em telemóveis ou tablets através de um browser, uma aplicação móvel poderia fornecer um conjunto extra de funcionalidades ao utilizador (por exemplo: utilização online, manutenção de um registo de avistamentos no dispositivo). O conceito foi concretizado para apenas um dos modelos do Biolens, no caso o Lepilens, com o desenvolvimento de uma aplicação para iOS (FIGURA 5).
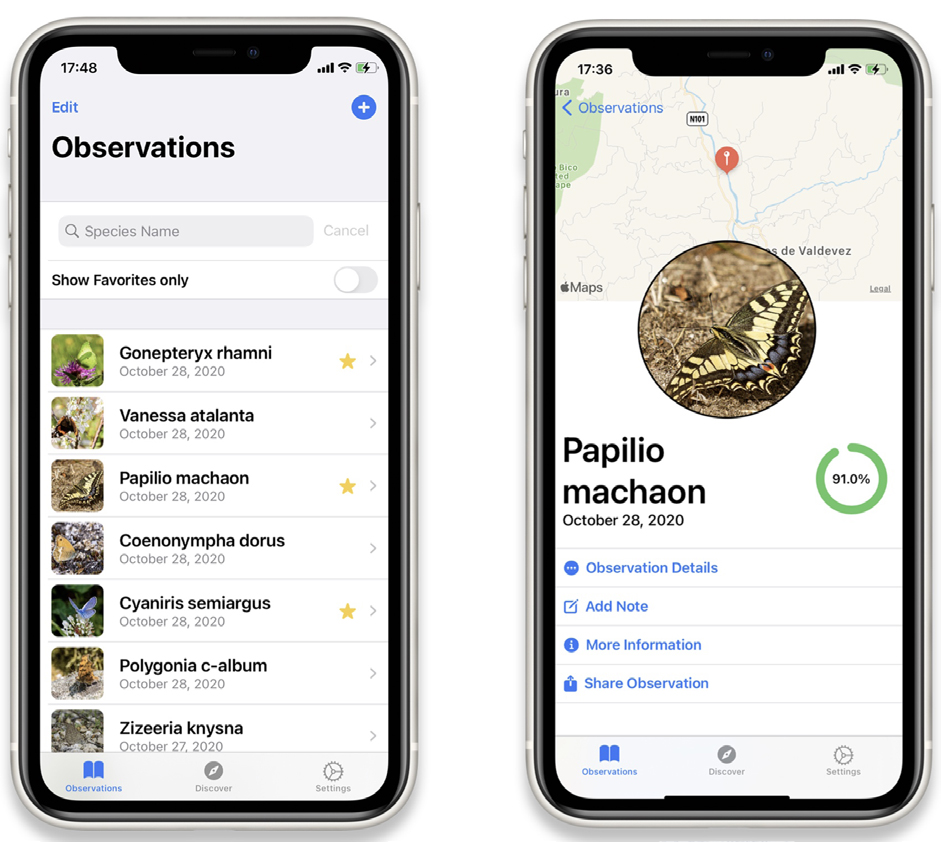
A aplicação permite que fotografias obtidas com o telemóvel ou carregadas de outros dispositivos ou da Web, possam ser identificadas pelo modelo Lepilens. Ao contrário da aplicação Web, a aplicação iOS funciona sem necessidade de ligação à Internet, uma situação comum para quem realiza trabalho de campo. Assim sendo, todos os avistamentos de borboletas são documentados, com data e hora, coordenadas GPS do local e notas opcionais do utilizador, e guardados numa base de dados SQLite3. Uma vez ligado à internet, o utilizador poderá optar por descarregar esses dados para o seu próprio computador ou contribuir com eles para uma plataforma existente, por exemplo, o iNaturalist ou o Observation.org.
A arquitetura da aplicação móvel permite que seja facilmente estendida para incluir os restantes modelos suportados pelo Biolens. Este trabalho, ainda que menos criativo e mais sistemático, faz parte dos planos futuros de desenvolvimento do projeto.
Agradecimentos
Os autores gostariam de expressar o seu agradecimento pela preciosa ajuda recebida dos especialistas Eduardo Marabuto, Pedro Pires e Ernestino Maravalhas (grupo Facebook “Lepidoptera (Borboletas) em Portugal”), Albano Soares (grupo Facebook “Libélulas e Libelinhas de Portugal”), Miguel Porto (Sociedade Portuguesa de Botânica) e Henrique Alves (Parque Biológico de Gaia). Os recursos computacionais da Google Cloud utilizados foram parcialmente disponibilizados ao abrigo da iniciativa Google Cloud Research Credits Program.
Referências
- 1 LOPES, L. B. et al., Biolens, DCC/FCUP.
- 2 MAMEDE, T., On using Deep Learning for Automatic Taxonomic Identification of Butteries, UC de Projecto, Lic. Ciência de Computadores. 2020.
- 3 MAMEDE, T., Lepilens iOS app, Apple Store. 2021.
- 4 FILGUEIRAS, A., FloraLens: a deep learning model for portuguese ora, Dissertação de mestrado. 2022.
- 5 MARQUES, M., A Portuguese Flora Identification Tool Using Deep Learning, Dissertação de mestrado. 2020.
- 6 Google Cloud, AutoML Vision Documentation.
- 7 ABADI, M. et al., TensorFlow: a system for large scale machine learning, USENIX, 265—283. 2016.
- 8 TAN, M. et al., Mnasnet: Platform-aware neural architecture search for mobile, IEEE, 2820—2828. 2019.
- 9 INATURALIST CONTRIBUTORS, iNaturalist Research-grade Observations, GBIF.org.
- 10 VRIES, H. & LEMMENS, M., Nature data from around the World, GBIF.org.
- 11 OUARD, A. A. et al., Pl@ntNet observations, GBIF.org.
- 12 SOCIEDADE PORTUGUESA DE BOTÂNICA, Flora-On: Flora de Portugal Interactiva.
- 13 PIRES, P., Lusoborboletas: borboletas de Portugal.
Este artigo já foi visualizado 5680 vezes.



