UMG-SP2
📧 , 📧 , 📧 , 📧
- * Universidade de Lisboa
- ɫ Universidade do Porto
- ‡ Universidade do Porto
- + Universidade do Porto
Referência Rodrigues, J. F., Ramos, M. J., Paiva, P., Fernandes, P. A., (2024) UMG-SP2, Rev. Ciência Elem., V12(3):032
DOI http://doi.org/10.24927/rce2024.032
Palavras-chave
Resumo
A imagem «UMG-SP2» foi submetida por Maria João Ramos, Pedro Paiva e Pedro A. Fernandes, e está disponível no Banco de Imagens da Casa das Ciências.
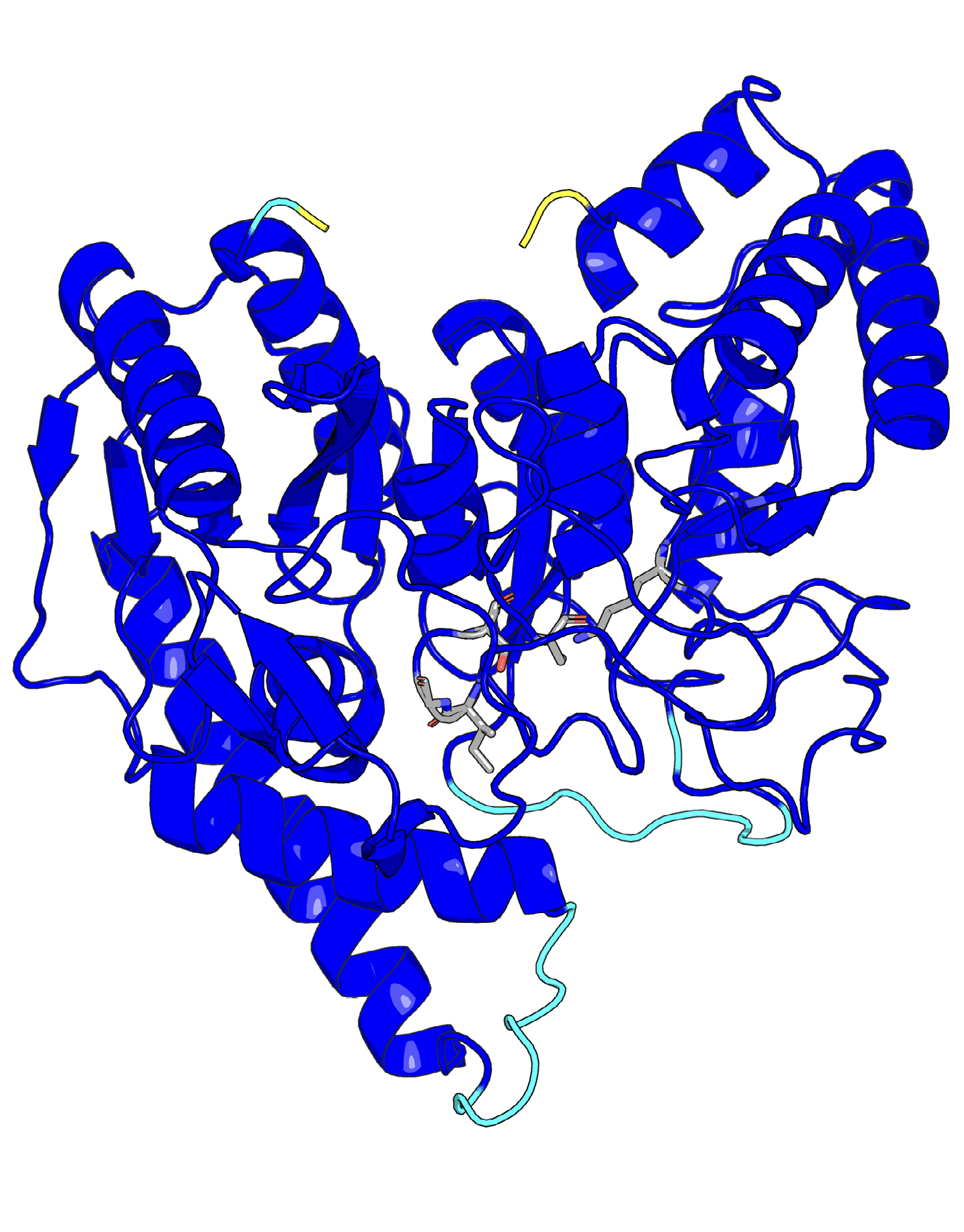
A figura apresenta a previsão da estrutura tridimensional proteica da uretanase UMG-SP2, gerada pelo software AlphaFold2, cujos autores foram distinguidos com o Prémio Nobel da Química de 2024. Esta enzima demonstrou grande capacidade para degradar a ligação uretano dos plásticos poliuretanos, revelando elevado potencial para um processo de reciclagem eficiente e ecologicamente amigável, baseado na despolimerização enzimática. No entanto, há ainda margem para melhorar a sua eficiência. O objetivo é atingir uma taxa de reciclagem em que uma enzima consiga degradar, pelo menos, 90% do plástico poliuretano em menos de 24 horas. Aumentar a eficiência catalítica das uretanases pode ser a chave para tornar o processo de reciclagem ecológica de plásticos mais eficaz. Neste contexto, o design computacional de proteínas, desenvolvido por muitos incluindo o cientista que compartilhou o Prémio Nobel da Química de 2024, pode contribuir significativamente para melhorar a eficiência enzimática na despolimerização enzimática, nomeadamente a da enzima UMG-SP2.
Pedro Paiva | Pedro Fernandes | Maria João Ramos
Universidade do Porto
A visualização da estrutura da proteína uretanase UMG-SP2 gerada pelo AlphaFold2 é muito interessante numa perspetiva didática e artística. As dobras e contornos intrincados da proteína criam uma interação dinâmica de formas e cores, que faz lembrar uma escultura moderna de serpentinas de papel azul. A utilização de espirais justapostas a ligações coloridas tridimensionais, chama a atenção do observador para as caraterísticas únicas da proteína.
Além disso, a representação dos centros ativos e das locais de ligação da proteína pode ser comparada a uma escultura abstrata e convida à interpretação e à exploração da complexidade molecular. A clareza e precisão das previsões do AlphaFold2 não só servem objetivos científicos, como também evocam a beleza da arquitetura molecular. Nesta perspetiva, esta visualização faz uma ponte entre ciência e arte, mostrando como a tecnologia avançada pode iluminar a elegância das estruturas bioquímicas complexas.
José Francisco Rodrigues
com ajuda do ChatGPT e DeepL
Universidade de Lisboa
Este artigo já foi visualizado 744 vezes.



